Description
Beschreibung der Experimente
1) Chemische Fällung von Proteinen aus der Kuhmilch (alle Gruppen, 30 min)
Material:
- Falcon-Schraubdeckelgefäße
- Kuhmilch 3,5% Fettgehalt
- Lactosefreie Milch
- Essigsäure konzentriert
- Ethanol absolut
- Zentrifuge
Durchführung: Je zwei SuS werden im Experimentallabor eine Milchprobe zu 50 ml erhalten und entweder mit Essigsäure oder Ethanol die Milchproteine fällen. Die Suspensionen werden anschließend zentrifugiert, um die präzipitierten Proteine zu pelletieren. Im Plenum werden die erhaltenen Ergebnisse diskutiert.
Anschließend folgt ein Impulsvortrag zur Zusammensetzung von Kuhmilch mit dem Schwerpunkt auf die enthaltenen Proteine und den Milchzucker (Lactose). Damit wird auf die Phase des Gruppenpuzzles überführt. Hierfür werden die SuS in drei Gruppen aufgeteilt.
2a) Expertengruppe Proteinbestimmung (2,5 h)
Material:
- Bradford-Stammlösung
- Eppendorf-Reaktionsgefäße mit BSA-Eichlösungen, mit Proben der Kuhmilch und der laktosefreien Milch, Wasser als Referenz
- Halbmikroküvetten aus Polystyrol
- Vis-Spektrometer
- Pipetten: 1000 µl & 20 µl
- Pipettenspitzen blau, gelb
- Verschiedenes: Abfalleimer, Edding, Küvettenständer, Eppiständer
- Computer mit Excel-Installation zur Auswertung
Durchführung: Ansetzen der Bradford-Reaktion mit definierten BSA-Lösungen, Messen der Absorbanz der inkubierten Proben bei 595 nm, Eintragen der Messwerte in das vorbereitete Excel-Spreadsheet, Erstellen der Kalibrationsgerade.
Wiederholung der Bradfordreaktion mit den Proben aus der Kuhmilch und der laktosefreien Milch, Bestimmung der Proteinkonzentration mithilfe der erstellten Kalibrationsgerade.

Abbildung 1: Links: Farbverlauf der Bradfordreaktion in den verwendeten Halbmikroküvetten. Rechts:
Das verwendete Vis-Spektralphotometer
2b) Expertengruppe SDS-Polyacrylamidgelelektrophorese (SDS-PAGE) (2,5 h)
Material:
- Vorbereitete SDS-Gele (Assistenten)
- SDS-Laufkammern
- SDS-Laufpuffer nach Laemmli
- SDS-Probenpuffer nach Laemmli
- Power Supply für die SDS-PAGE mit Anschlusskabeln
- Eppendorf-Reaktionsgefäße
- Pipetten: 20 µl & 2,5 µl
- Pipettenspitzen gelb und weiß
- Geldokumentationsgerät
- Heizblock
- Färbelösung (Coomassie Brilliant Blue, EtOH, Essigsäure)
- Entfärbelösung (Essigsäure, EtOH)
- Verschiedenes: Abfalleimer, Edding, Eppiständer
Durchführung: Versetzen der Proben der Milch aus Versuch 1 (unbehandelt, Pellets und Überstande der Denaturierungen), sowie der laktosefreien Milch in Eppis mit Laemmli-Probenpuffer. Erhitzen der Proben für 1 min bei 95 °C. Einspannen eines vorgegossenen SDS-Geles in die Laufkammer, Befüllen der Kathoden- und Anodenreservoirs mit SDS-Laufpuffer. Laden der Proben in die Probentaschen des SDS-Gels, zusätzlich einen Größenstandard und eine Probe der Lactase (aus Versuch 2c) als Referenzen, Start der elektrophoretischen Trennung.
Nach Ende der Trennung Färben des SDS-Gels mit Coomassie Brilliant Blue, dann Entfärbung des Hintergrunds und Dokumentation des Gels.
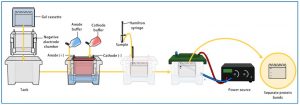
Abbildung 2: Schematischer Ablauf einer SDS-PAGE. Quelle: Wikipedia, http://en.wikipedia.org/wiki/SDS-PAGE
2c) Expertengruppe Enzymkinetik (2,5 h)
- Material:
- Eppendorf-Reaktionsgefäße
- Pipetten: 20 µl, 200 µl & 1000 µl
- Pipettenspitzen blau, gelb, weiß
- Eppendorf-Reaktionsgefäße
- UV/vis-Spektrometer mit PC
- PC mit Software (Excel)
- Halbmikroküvetten aus Polystyrol
- Lactase (ß-Galactosidase)
- 5 mg/ml Stammlösung des Substratanalogons ortho-Nitrophenylgalactosid (o-NPG)
- 50 mM Natrium-Kalium(Na-K)-Phosphatpuffer-Lösung pH 6,8
- 50 mM MgCl2-Lösung
- Eisbad mit Eis
- Verschiedenes: Abfalleimer, Edding, Küvettenständer, Eppiständer
Durchführung: Ansetzen der Reaktionen (ohne Enzym) auf Eis mit ansteigenden Konzentrationen an
o-NPG. Start der Reaktionen jeweils durch Enzymzugabe und Messen des Absorptionsverlaufs bei 420
nm über die Zeit. Infolge des o-NPG-Abbaus sinkt die Absorbanz, so dass die enzymatische Reaktion
optisch verfolgt werden kann. Bestimmung der Reaktionsgeschwindigkeit aus der Steigung für jede
Reaktion unter Einbezug des molaren Extinktionskoeffizienten von o-NPG bei 420 nm. Doppelt-
reziprokes Auftragen (1/v gegen 1/[S]) aller erhaltenen Werte für die Reaktionsgeschwindigkeit und
die zugehörige Substratkonzentration in einem Lineweaver-Burk-Plot in Excel. Ermittlung der
Michaelis-Menten-Konstante KM und der maximalen Reaktionsgeschwindigkeit vmax.
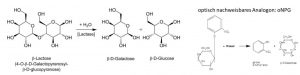
Abbildung 3: Reaktionen der Lactase. Links: Reaktionsschema des Lactoseabbaus zu β-D-Galactose und β-D-Glucose durch
Lactase. Rechts: Reaktionsschema des Abbaus von ortho-Nitrophenylgalactosid zu 2-Nitrophenol (gelbes Produkt) und β-D–
Galactose durch die Lactase.
3) Bildung der Puzzlegruppen und Ergebnisaustausch (30 min)
Es werden nun Puzzlegruppen mit je eine/r Schüler:in aus jeder Expertengruppe gebildet und die
Ergebnisse in diesen Gruppen vorgestellt und diskutiert. Die Gruppe 2a stellt den Proteingehalt in der
Kuhmilch und der laktosefreien Milch vor und erläutert das Nachweisverfahren. Gruppe 2b teilt ihre
Analyse der Proteinzusammensetzung mit den anderen Gruppen. Dabei fällt auf, dass die Lactase in
Milch nicht enthalten ist, in der laktosefreien Milch aber möglicherweise schon (von der industriellen
Behandlung abhängig). Gruppe 2c teilt ihre Ergebnisse zur Enzymkinetik der Lactase, die den
Milchzucker abbauen kann. In der Synthese erarbeiten sich die SuS die Erkenntnis, dass die Lactase
den Milchzucker Lactose effizient abbauen kann, in der Milch selbst aber natürlich nicht enthalten ist.
Weiterhin lernen sie damit, dass laktosefreie Milch durch Behandlung mit Lactase hergestellt wird.
4) Konsolidierung durch PowerPoint-Vortrag und Diskussion (30 min)
Die Lehrperson fasst die Ergebnisse in einem interaktiven PowerPoint-Vortrag zusammen und stellt
die Methoden der Cryo-Elektronenmikroskopie und der Röntgenkristallographie zur
Strukturbestimmung von Proteinen kurz vor. Dabei werden die Strukturelemente von Proteinen und
das Konzept der Elektronendichte vorgestellt. Im Gespräch werden die Lernerfolge gesichert.